Liganden-Entwicklung
Entwicklung glykosylierter Impfstoffproteine
Uropathogene E. coli-Stämme verursachen vor allem bei der stationären Behandlung anderer Erkrankungen häufig schwerwiegende Harnwegsinfektionen. Neben der zusätzlichen gesundheitlichen Belastung für die Patient*innen, werden dadurch auch erhebliche zusätzliche Behandlungs- und Folgekosten generiert.
Ziel des Projekts ist die Entwicklung eines Impfstoffs zur Stärkung der Immunantwort gegen Harnwegsinfektionen, insbesondere von Klinikpatient*innen. Dabei soll die Immunantwort gezielt gegen das für die Besiedlung der Harnwege essentielle bakterielle Protein YghJ gerichtet werden.
Forschende des dänischen Unternehmens GlyProVac LLC. haben herausgefunden, dass Bakterien wie E. coli einen Großteil ihrer Proteine mit Zuckermolekülen (Glykosylierung) modifizieren. Diese Modifikationen haben einen signifikanten Einfluss auf die Immunantwort und sind somit auch für die Entwicklung schützender Impfstoffe relevant. GlyProVac LLC. hat Verfahren entwickelt, um diese Modifikationen nachzuweisen und zu analysieren. Im Rahmen des Projektes werden nun spezielle Wirtsstämme für Herstellung rekombinanter Proteine entwickelt, die dem natürlich vorkommenden YghJ-Protein sehr nahe sind, jedoch eine verbesserte Immunantwort induzieren.
Um die Immunantwort auf glykosylierte und nicht-glykosylierte YghJ-Impfstoffproteine detailliert zu studieren und mit der Immunantwort auf das natürliche Antigen zu vergleichen, müssen die vom Immunsystem gebildeten Antikörper präzise analysiert werden. Bisher verfügbare Methoden erwiesen sich dafür jedoch als unzureichend.
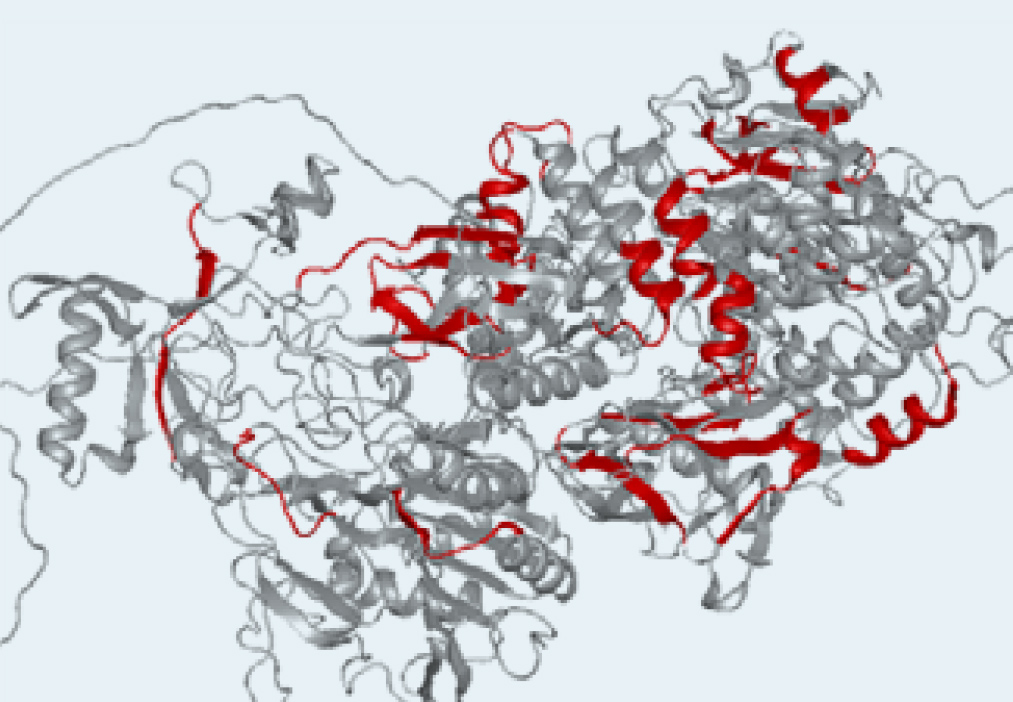
Forschende des Fraunhofer IZI und der epitopic GmbH entwickelten deshalb ein Verfahren zur schnellen und präzisen Identifizierung und Analyse von Epitopen, den molekularen Strukturen die von Antikörpern erkannt und gebunden werden. Damit konnten mehr als 20 Epitope des YghJ-Proteins identifiziert werden, welche zur Bewertung der Impfstoffkandidaten herangezogen werden können (siehe Abbildung). Die weitere Verbesserung der Methode soll es zudem ermöglichen, auch Strukturen zu finden, die gegen die Glykosylierung selbst gerichtet sind.
Vergleiche zwischen Tiermodell und Seren von Patient*innen konnten bereits bestätigen, dass die entwickelten Impfstoffkandidaten eine Immunantwort hervorrufen, die der natürlichen Reaktion gleicht. Die im Rahmen des Projektes entwickelten Nachweisverfahren, werden auch für die spätere klinische Entwicklung wichtig sein.
Partner
GlyProVac LLC.; epitopic GmbH; Odense University Hospital; Klinikum St. Georg Leipzig



Biosensoren für den Nachweis von Infektionserregern
Die Coronapandemie hat deutlich gemacht, wie wichtig robuste Technologien zur schnellen und akkuraten Analyse von Krankheitserregern und Antikörpern sind, um Infektionsausbrüche und Immunität in größeren Bevölkerungsgruppen zu überwachen. Ziel des Projekts ist die Entwicklung einer immundiagnostischen Technologieplattform, welche flexibel an ein dynamisches Pandemiegeschehen adaptierbar ist.
Zentrale Elemente der Entwicklung sind Elektroden aus Bor-dotiertem Diamant (BDD), die mit Epitopen, das sind vom Antikörper erkannte, im Erreger vorhandene Strukturen, zu sensitiven und effizienten Biosensoren kombiniert werden.
Als Musteranwendung wurde zunächst die spezifische Detektion von SARS-CoV-2 adressiert. Das Fraunhofer IZI bringt hierbei seine Kompetenzen im Bereich der Identifizierung und Entwicklung von peptidbasierten Epitopen ein, auf deren Grundlage sensible und spezifische Nachweissysteme entwickelt werden. Verwendet werden bereits existierende Epitope aus Arbeiten mit hunderten von Patient*innenseren. Im Rahmen des Projektes wurden dazu auch Seren Omikron-infizierter COVID-19-Patient*innen verwendet. Gleichzeitig wurden in Hinblick auf Long-COVID Seren und Daten von Patient*innen auf potenzielle Epitope von Auto-Antigenen gegen körpereigene Proteine ausgewertet, denen eine wichtige Rolle bei der Entwicklung von Long COVID zugesprochen wird.
Gemäß einem Baukasten Prinzip, soll die Plattform flexibel und schnell an unterschiedliche Krankheitserreger oder immunologisch relevante Fragestellungen angepasst werden können. Ziel ist es, zukünftig innerhalb weniger Wochen nach einem Infektionsausbruch Geräte zur Point-of-Care-Diagnostik, zum schnellen und kosteneffizienten Monitoring von Infektionskrankheiten und zur Bestimmung des Immunstatus geimpfter oder genesener Personen verfügbar zu machen.
Partner
Fraunhofer USA Center Midwest CMW, Coatings and Diamond Divison; Fraunhofer-Institut für Zuverlässigkeit und Mikrointegration IZM
EpiCoV-2020: Identifizierung von Antikörper-Epitopen von SARS-CoV-2 und endemischen Viren
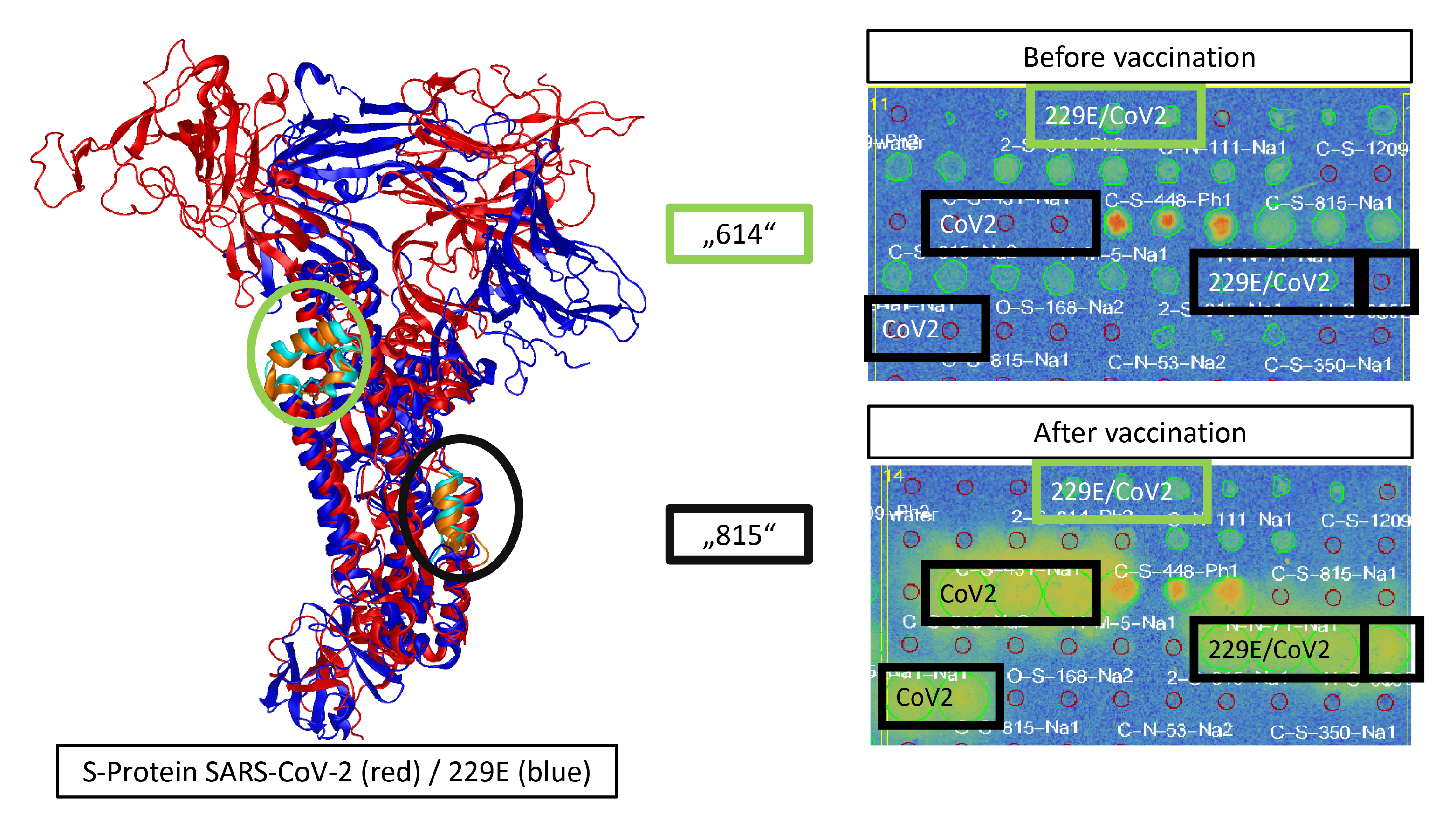
Coronaviren begleiten die Säugetiere schon sehr lange Zeit. Mindestens vier verschiedene endemische Coronaviren zirkulieren in Menschen und haben ihre Spuren im Immunsystem hinterlassen. Daher zielen diese Arbeiten nicht allein auf die Identifizierung von Epitopen, die von Antikörpern in SARS-CoV-2 erkannt werden, sondern auch auf ihre endemischen Verwandten. Mit der »Epitop-Fingerprinting«-Methode können auch kleine Unterschiede der von Antikörpern erkannten Aminosäuresequenz der teilweise sehr ähnlichen Virusproteine berücksichtigt werden. In der Tat fanden die Analysen neben SARS-CoV-2-spezifischen Epitopen auch sehr ähnliche Antikörper in Seren nach Infektion mit Erkältungskrankheiten von vor 2020, aktuellen COVID-19-Infektionen und nach Impfung. Eine große Zahl solcher Epitop-Peptide wurde synthetisiert, mit einer speziellen Technik als Arrays auf Objektträger gedruckt und mit über 1 000 Serumproben getestet. An Peptid-Spots gebundene Serumantikörper konnten über eine Fluoreszenz-Markierung, anschließendes Scannen im Laserscanner und mittels einer im Rahmen des Projekts neu entwickelten robusten automatisierten Analyse der erhaltenen Bilder auch quantitativ sehr einfach erfasst und ausgewertet werden. Nur dank dieser Entwicklung konnte eine so große Zahl von Messungen rasch und zuverlässig ausgewertet werden.
Durch Korrelation der Ergebnisse von allen drei verschiedenen klinischen Gruppen wurden zahlreiche Antikörper gegen das S-Protein und die RNA-Polymerase gefunden, die ihren Ursprung in früheren Infektionen mit endemischen Viren haben und teilweise schon vor der Impfung bzw. unmittelbar nach der SARS-CoV-2-Infektion im Serum zirkulieren. Überraschenderweise erkennen einige kreuzreagierende Antikörper auch Epitope im S-Protein, die sich zwar in der Sequenz aber kaum in der Struktur unterscheiden (Abb. 1).
Diese und andere Epitope können in der Zukunft helfen, nicht nur global Antikörper an einem Protein, sondern auch spezifisch einzelne Antikörper und damit die individuell sehr verschiedenen Immunantworten auf COVID-19 und Impfstoffe nachzuverfolgen.

CoV-tot – Untersuchung des Einflusses einer Virusinaktivierung auf das Epitop-Spektrum von (COVID-19)-Seren
Serologische Diagnostik für COVID-19 wird gegenwärtig nur auf Basis von Proteinen angeboten. Neben überraschend falsch positiven Ergebnissen werden aus der klinischen Diagnostik vor allem Probleme durch Vorerkrankungen mit verwandten Coronaviren vermeldet. Die Erkennungsstellen der Patient*innenantikörper (Epitope) sind nämlich nur teilweise spezifisch für SARS-CoV-2, während sich andere auf vielen verwandten Coronaviren wiederfinden.
Es war daher notwendig, die serologischen Tests auch auf Basis von definierten Epitopen von SARS-CoV-2 bzw. anderen Coronaviren aufzubauen, die mit Hilfe verschiedener spezifischer und ubiquitärer Epitope eine ebenso einfache wie hochgradig individualisierte Diagnostik erlauben. Am Fraunhofer IZI verfügt die Arbeitsgruppe Liganden-Entwicklung über umfangreiche Erfahrungen mit der Identifizierung von Epitopen direkt aus Seren. Es zeigt sich bereits in dieser Studie, dass es bei SARS-CoV-2-Infektionen zu einer stark personalisierten Immunantwort geprägt durch frühere Corona-Infektionen kommt.
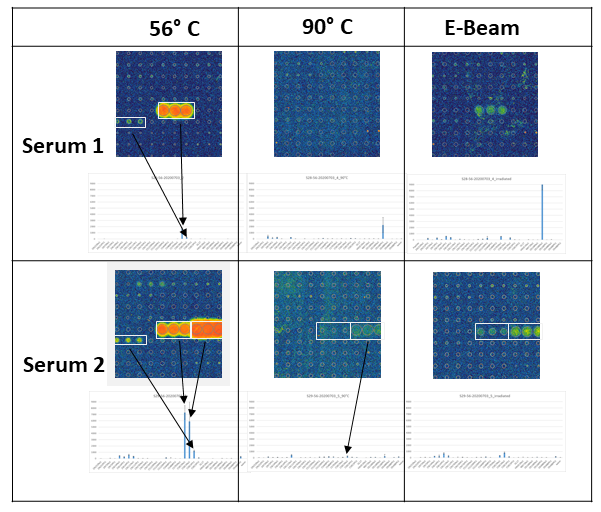
Bei allen diagnostischen Arbeiten werden die Seren aus infizierten Patienten*innen aber zur Virusinaktivierung vorbehandelt, bevor sie in den Tests eingesetzt werden können. Im Rahmen von CoV-tot konnte nachgewiesen werden, dass verschiedene Methoden zur Virusinaktivierung einen sehr unterschiedlichen Einfluss auf verschiedene Antikörper in den Seren haben. Dies kann im Einzelfall zu ganz unterschiedlichen Ergebnissen bei serologischen Untersuchungen führen. Darüber hinaus haben die Studien ergeben, dass eine leichte Denaturierung der Probe die Blockierung einzelner Antikörper durch unbekannte Serumbestandteile aufzuheben scheint.
Dadurch können bessere Ergebnisse erzielt werden. Zwei Methoden der Virusinaktivierung konnten als geeignet identifiziert werden, zum einen die Erhitzung auf 56 Grad Celsius für zehn Minuten bei flüssigen Proben und zum anderen die Behandlung mit 70 % Ethanol, wenn die Antikörper immobilisiert an Protein A vorliegen.
Die im Rahmen von CoV-tot gewonnenen Erkenntnisse waren ein Startpunkt zur Erforschung von Unterschieden im Krankheitsverlauf auch in Zusammenhang mit klinischen Studien zur medikamentösen Behandlung von COVID-19.
Partner
Klinikum St. Georg, Leipzig
Förderung
Gefördert im Rahmen der Internen Programme der Fraunhofer-Gesellschaft, Fördernummer Anti-Corona 131-600034.
Mapping von Allergen-Epitopen in Seren
Die Immundiagnostik von Krankheiten beruht zurzeit in der Regel auf Proteinen oder Extrakten, die direkt aus den pathogenen Organismen oder biotechnologisch hergestellt werden. Der Nachteil ist, dass Varianten, wie sie zum Beispiel bei Grippeviren vorliegen, nur schwer unterschieden werden können. Es konnten Verfahren zur exakten Identifikation der Bindungsstellen von Patient*innen-Antikörpern (Epitopen) entwickelt werden, die auch direkt in Seren Anwendung finden können. Das erlaubt die zuverlässige Identifizierung des Erregers, des ursächlichen Antigens bei einer Allergie oder vielen Indikationen wie (Auto)Immun- und Infektionskrankheiten sowie neue Therapie- und Forschungsansätze.
Lebensmittelallergien sind seit vielen Jahren ein Forschungsschwerpunkt der Arbeitsgruppe Liganden-Entwicklung. Eine stetige Zunahme gerade dieser Erkrankungen ist in den letzten Jahren zu beobachten. Diagnosen mit Haut-Pricktest sind nur begrenzt hilfreich, da vor allem viele Pflanzenproteine sehr ähnlich in ihrem Aufbau sind. Eine epitopbasierte Diagnostik ist wahrscheinlich die einzige Alternative zu aufwendigen klinischen Untersuchungen. Diese setzen in der Regel eine Blutentnahme voraus, wobei einzig die Provokation mit den Lebensmitteln, die stationär und unter ärztlicher Beobachtung in einer Klinik durchgeführt werden muss, bisher als sicherer Beweis einer Allergie gilt. Eine effiziente Diagnose, geeignete Behandlung und Anpassung der Ernährung ist daher bei vielen Patient*innen kaum möglich.
In einem ersten Projekt am Beispiel der Sojaallergie wurde gezeigt, dass tatsächlich einige wenige Epitope zur sicheren Identifikation von sensibilisierten Personen und auch solchen mit klinischen Symptomen ausreichen könnten. Diese Peptide sollen zukünfig in einem einfachen Test zum Einsatz kommen, der entsprechende Antikörper auch in einem Blutstropfen nachweisen könnte. Dies wäre ebenso ein Modell für Tests auf Infektionskrankheiten, die Effizienz von Impfstoffen oder Autoimmunerkrankungen.
Ein besonders umfangreiches Projekt wird von der Fraunhofer-Zukunftsstiftung gefördert, das auch national und international von Allergolog*innen mit großem Interesse verfolgt wird. In Kooperation mit mehreren anderen Fraunhofer-Instituten und Kliniken wird im Projekt FoodAllergen an einem holistischen Ansatz zum Umgang mit Lebensmittelallergien geforscht. Dieser umfasst auch die Identifizierung der Allergene in Lebensmitteln sowie neue Verfahren zu Herstellung von Lebensmittelzutaten mit reduziertem allergenen Potenzial. Es wurden inzwischen die Epitope für verschiedenste Pflanzenallergene in Nahrungsmitteln identifiziert. Eine Umsetzung in Tests für Patient*innen ist in Vorbereitung.
Epitop / Mimotop Mapping
Peptid Phage Display Bibliotheken von der Größe, wie sie am Fraunhofer IZI zur Verfügung stehen, erlauben eine rasche und zuverlässige Identifizierung von Epitopen und Mimotopen sowohl von monoklonalen als auch polyklonalen Antikörpern. Die gefundenen Peptide liefern nicht nur wertvolle Information zur Bindestelle am Protein sondern sie können auch in serologischen Assays und zur Reinigung von rekombinanten Antikörpern oder spezifischen Antikörpern aus polyklonalen Mischungen verwendet werden.
Diese Routinearbeit wird wegen ihrer kurzen Bearbeitungszeit und leicht überprüfbaren Ergebnissen gern von Firmen im Rahmen der Entwicklung von Antikörpern gewünscht.
ZellFix – Oberflächenaktivierung besser als Plasma?
In einem gemeinsamen Projekt mit dem Fraunhofer IFAM wurde entdeckt, dass eine einfache, mit zellbiologischen Verfahren kompatible Enzymreaktion genutzt werden kann, um Peptidliganden kovalent an ihre Rezeptoren auf Zellen zu binden. Daraufhin initiierten beide Institute das von der Fraunhofer-Gesellschaft geförderte Projekt ZELLFIX, um die gleiche Reaktion zur Modifikation von Oberflächen einzusetzen.
Zellen haften ungern auf den Polymeren von Kulturschalen. Normalerweise werden Polystyroloberflächen mit Plasma behandelt, was aber auch zu unerwünschten, teilweise toxischen Nebenprodukten führt. Mit dem neuen Verfahren könnten zum einen Peptid- und Proteinliganden die Anheftung der Zellen verbessern und zum anderen einzelne Oberflächenproteine kovalent direkt an die Platten gebunden werden, wie dieses auch auf plasmaaktivierten Oberflächen geschieht.
Im Projekt ZELLFIX wurden zunächst die ursprünglichen Annahmen experimentell bestätigt. Sogar abgegrenzte Bereiche von Zellkulturschalen lassen sich mit geringem Aufwand so modifizieren, dass Zellen nur dort anhaften. Diese Methode ist auch geeignet, um die lokale Anheftung von Proteinen selbst bei handelsüblichen, bereits plasmaaktivierten, Oberflächen zu verbessern.
Diese Verfahren werden bereits mit Partnern aus Industrie und Fraunhofer-Gesellschaft für erste spezielle Anwendungen erprobt. Zudem konnten über den biologischen und medizinischen Bereich hinaus zusätzlich neue Anwendungen identifiziert werden.

